Vor zwei Wochen nahm ich an einer Diskussionsrunde zu personalisierten genetischen Tests Teil. Firmen wie 23andMe bieten für ein paar Hundert Dollar an, die eigene DNA zu analysieren und dann Rückschlüsse auf Krankheitsrisiken, aber auch auf die persönliche Abstammung zu ziehen. Ich habe hier im Blog von der Diskussionsrunde (live) berichtet.
Im Zuge meiner Vorbereitungen habe ich aktuelle Angebote für diese personalisierten genetischen Tests recherchiert. 23andMe testet derzeit für 300 Dollar. Das sind 100 Dollar weniger als vor knapp drei Jahren. Günstiger ist natürlich gut, und auch die Zahl der ausgewerteten Merkmale ist von rund 120 vor drei Jahren auf derzeit 243 gestiegen. Insgesamt werden dafür von 23andMe Daten von rund einer Million SNPa analysiert. Die Technik, die 23andMe verwendet nennt sich Genotypisierung. Dabei werden Mutationen in kurzen DNA-Abschnitten (single nucleotide polymorphisms, SNPs), Wahrscheinlichkeiten für bestimmte Krankheiten zugeordnet. Die Wahrscheinlichkeiten wurden (und werden weiter) in sogenannten genomweiten Assoziationstudien (GWAS) bestimmt.
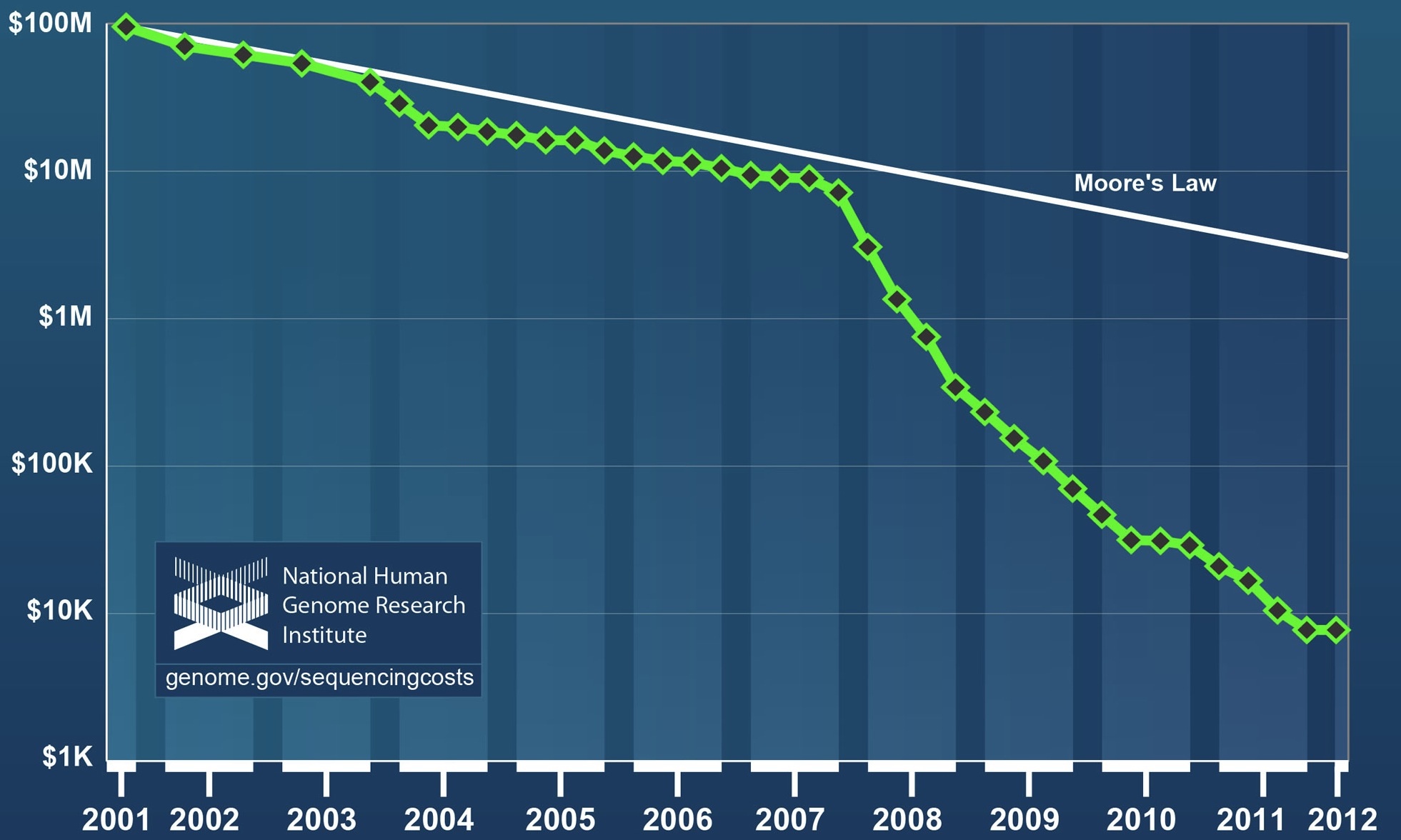
Die Genotypisierung ist nicht die einzige Möglichkeit, Daten zu Krankheitswahrscheinlichkeiten durch die Analyse der DNA zu gewinnen. Die Alternative ist die DNA Sequenzierung. Und hier sind die Kosten in den letzten Jahren dramatisch gefallen. Die Grafik des National Human Genome Research Institutes zeigt detailliert, wie sich die Kosten für die DNA Sequenzierung seit 2001 entwickelt haben. Der Preisverfall übeflügelt das Moorsche Gesetz weit und besonders auffällig ist der Preisknick nach 2007 (siehe Abbildung oben). Dieser hängt mit einer technischen Neuerung zusammen: Das sogenannte Next Generation Sequencing hat vor ein paar Jahren das traditionelle Sanger-Sequenzing in den großen Sequenzierzentren abgelöst. Das Next Generation Sequencing unterschiedet sich vor allem durch eine massive Parallelisierung der Sequenziervorgänge, hier ein Übersichtsartikel über Sequenziertechniken.
Weltweite Verteilung der Next Generation Sequenziermaschinen, Stand 09/2012. Quelle http://omicsmaps.comInteressant ist auch die weltweite Verteilung der Next-Generation Sequenziermaschinen, sie bildet annährend die Verteilung der Wissenschaftsausgaben ab. Aktuell sind 2035 dieser Sequenziergeräte im Einsatz. 922 davon stehen in Nordamerika, 604 in Europa und 377 in Asien. Das weltweit größte Sequenzierzentrum ist das BGI in China mit 166 Maschinen. In Deutschland stehen 142. Trotz dieser Explosion an Sequenzierpower kostet aktuell die Sequenzierung eines kompletten menschlichen Genoms noch deutlich zu viel, um kommerziell mit SNP-Genotyping konkurrieren zu können. Das sogenannte Exomesequening, bei nicht das ganze Genom sequenziert wird, sondern nur die Teile, die tatsächlich für Proteine kodieren, ist jedoch ein günstigerer Zwischenschritt, der mittelfristig die Genotypisierung wenn nicht ganz ablösen, doch zumindest ergänzen wird. 23andMe muss derzeit dennoch keine Angst vor Konkurrenz haben. Das Unternehmen bietet (für bestehende Kunden) die Sequenzierung des Exoms mit 80-facher Coverage bereits für 999 Dollar an.
Trotz aller Preisstürze: Deutlich günstiger und häufig zuverlässiger als genetische Tests zur Vorhersage von Krankheiten sind Blutdruck messen, ein Blick auf den Bauchumfang und auf persönliche Laster.
Quelle Bild oben: Wetterstrand KA. DNA Sequencing Costs: Data from the NHGRI Large-Scale Genome Sequencing Program www.genome.gov/sequencingcosts.
Übertragungsfehler?
Bitte wenn es geht den ersten Text löschen.
Will ja nicht maulen, aber der Satz….
Die Genotypisierung ist nicht die einzige Möglichkeit, Daten zu Krankheitswahrscheinlichkeiten durch die Analyse der DNA zu gewinnen. Die Alternative ist die DNA Sequenzierung.
Die Genotypisierung ist das molekularbiologische Pendant zur Phänotypisierung.
Beide beschreiben am Ende jedoch im Allgemeinen einen Phänotyp.
Welche Methode man hierfür einsetzt ist zunächst völlig egal.
SNP
s erhält man auch nur durch (DNA)Sequenzierung. Auch „Next Generation Sequencing“ ist eine Sequenzierung. In aller Regel werden SNPs eben durch das „technische Bindeglied“ der normalen Sanger-Sequenzierung und dem Next Generation Sequencing – dem Pyrosequencing – analysiert.Sanger-Sequenzierung –> Pyrosequencing-> 454 Pyrosequencing–Next Generation….
Man erhält einen Datensatz (egal wie) und verwendet diesen um auf einen Phänotyp zu schließen.
Diese „virtuelle“ Zuordnung zu einem Phänotyp nennt man Genotypisierung. Das Ergebnis der Genotypisierung ist am Ende sozusagen ein Phänotyp. Ohne vorliegen eines Phänotyps keine Genotypisierung.
z.B: genotypische Resistenztestung z.B.: bei HIV die Medikamenten-Resistenz. „Ich“ finde eine Mutation und ordne sie einer Resistenz zu. Die Resistenz gegenüber einem Medikament ist jedoch ein Phänotyp.
In deinem Beispiel wäre der Phänotyp: Erhöhte Wahrscheinlichkeit eine Krankheit x zu entwickeln.
Aveneer, du hast Recht, entschuldige die Ungenauigkeit
Ich habe auch deinen ersten Kommentar gelöscht. Spitze Klammern können zum Markup benutzt werden, sie funktionieren jedoch nicht als normale Buchstaben.