Schön, dass geistiges Eigentum in Deutschland einen so hohen Stellenwert genießt. Wörtliche Übernahmen einiger Textstücke und fehlende Quellenangaben in einer Dissertation einer 25 jährigen im Jahr 1980 führen dazu, dass eine gestandene Bundesministerin zurücktritt. Um Schaden von Amt, Ministerium, Partei und Regierung abzuwenden. Zu bewundern, die Rücktrittskultur in Deutschland, wie sie Kommentator Gustav schon letzte Woche ausgemacht hat.
Ich für meinen Teil finde es ja fast ein bisschen langweilig, dass die Geschichte so schnell vom Tisch ist. zu Guttenberg hat zur Freude aller zumindest ein paar Tage länger gekämpft, und in Relation zu anderen, aktuellen Fällen von Wissenschaftsbetrug ist die Causa Schavan, mit Verlaub, ein Fürzchen.
Zum Beispiel gibt es Diederik Stapel, ein 2011 suspendierter Professor der Sozialpsychologie an der Universität Tilburg und vormals in Groningen in den Niederlanden. Stapel ist der König der Wissenschaftsfälscher. Er hat einerseits existierende Datensätze so manipuliert, dass sie seinen Hypothesen entsprechen, oder die Rohdaten gleich komplett selbst erfunden. Er deckte diesen Vorgang durch scheinbar reguläre Vorbereitungen. Methoden und Studienbedingungen wurden mit Co-Autoren diskutiert und Fragebogen wurden entworfen, jedoch nie ausgeteilt.
Einer dieser Artikel hatte es sogar in Science geschafft. Inzwischen wurden 49 Artikel von Stapel zurückgezogen und wahrscheinlich wird die Zahl auf 65 Artikel ansteigen. Diederik Stapel ist geständig, die Hintergründe und Ergebnisse des Falles sind gut dokumentiert und können hier nachgelesen werden. Retraction Watch berichtet regelmäßig über frisch zurück gezogene Stapel-Artikel.
Sehr schön ist auch der Fall Anil Potti. Potti ist ausgebildeter Arzt und war bis 2010 an der Duke Universität in North Carolina beschäftigt. Er erforschte die Zusammenhänge von Genexpressionsänderungen, Krebs und Krebstherapie (Oncogenomics). Aktuell sind zehn seiner Artikel zurück gezogen, darunter Papers in bekannten Magazinen, wie NJEM, Nature Medicine und PNAS. Potti hat Microarraydaten im großen Stil gefälscht, deren Interpretation seinen Publikationen zu Grunde liegen. Retraction Watch verfolgt den Fall Potti ebenfalls, seit 2010.
Aktuell nimmt dieser Fall eine kuriose Wendung. Die Seite Retraction Watch wurde letzte Woche von Ihrem Provider Informiert, dass zehn der Blogartikel zu Anil Potti wegen Copyrightverletzungen nicht mehr online gestellt werden dürfen. Der Provider drohte weiter, dass bei wiederholten Copyrightverletzungen die gesamte Seite geschlossen werden könnte.
Kläger in diesem Copyrightfall war die Seite newsbulet.in, von der Adam Marcus und Ivan Oransky, die Macher von Retractionwatch, ihre Artikel zu Potti abgeschrieben haben sollten – und tatsächlich waren zehn Retractionswatchartikel identisch mit Artikel auf der indischen Seite (inzwischen dort gelöscht).
Ein eher technisches Detail legt nahe, dass die eigentlichen Urheber der Potti-Artikel die Autoren von Retractionwatch sind: Die Seite newsbulet.in wurde erst im Oktober 2012 registriert. Neun der zehn kopierten Artikel zu Potti wurden jedoch von Retractionwatch davor publiziert.
Potti, von dem zehn Artikel komplett zurück gezogen und etliche andere nachträglich geändert werden mussten hat übrigens laut Retractionwatch noch aktive Lizenzen in North Carolina und Missouri, kann dort also als Arzt praktizieren.
Kategorie: Medizin
-

Extreme Fälle von Betrug in der Wissenschaft
Verwandte Artikel im Blog: -
Poliowildviren im Abwasser in Ägypten
Gastartikel von Sabine Diedrich und Reinhard Burger
Im Großraum Kairo sind bei Routineuntersuchungen des Abwassers in Proben aus zwei verschiedenen Orten eingeschleppte Poliowildviren aus Pakistan nachgewiesen worden. Aus diesem Grund reagierten die ägyptische Regierung und die Global Polio Eradication Initiative (angeführt von der WHO, UNICEF, Rotary International und der amerikanische Gesundheitsbehörde) unmittelbar aus Vorsorge. Untersuchungen nach eventuellen Poliofällen starteten sofort, jedoch bis jetzt gibt es noch keinen Beweis, dass sich die eingeschleppten Viren verbreitet haben.
Bisher wurden zwar keine Fälle von Kinderlähmung in Ägypten gemeldet. Da jedoch nur etwa eines von 100 infizierten Kindern symptomatisch erkrankt, ist es schwer abzuschätzen, wie viele Personen betroffen sind.
Ägypten ist seit 2004 poliofrei. Die Weltgesundheitsorganisation plant nun eine Impfkampagne, bei der alle Kinder unter 5 Jahren in Ägypten geimpft werden sollen. Diese könnte sich jedoch unter den derzeitigen politischen Unruhen als sehr schwierig erweisen.
Pakistan ist neben Afghanistan und Nigeria eines der letzten drei Länder, in denen Poliowildviren endemisch zirkulieren. Nachdem dort Ende 2012 mehrere Impfhelfer ermordet wurden, mussten die Impfkampagnen teilweise gestoppt werden. Jedoch wurden die Impfkampagnen unter erhöhter Sicherheit in 29 Distrikten wieder aufgenommen mit dem Ziel, 13 Millionen Kinder zu erreichen. Die Zivilgesellschaft, religiöse Führer, Politiker und die Regierung bemühen sich sehr, die Impfkampagnen zu unterstützen.
Der Nachweis von Poliowildviren in bereits poliofreien Ländern verdeutlicht einmal mehr die hohe Gefahr der Wiedereinschleppung dieser Viren. Die im Robert Koch-Institut ermittelten Daten zeigen eine sehr gute Immunität der deutschen Bevölkerung gegen die drei Polioviren, so dass ein Polioausbruch bei uns eher als unwahrscheinlich anzusehen ist.
Jedoch zeigten die teilweise dramatischen Verläufe vergangener Ausbrüche nach Wiedereinschleppung, z. B. in der Republik Kongo oder Tadjikistan, das hohe Risiko für nicht geimpfte Bevölkerungsgruppen, an Polio zu erkranken. Diese Ausbrüche und die Polio-Nachrichten aus Ägypten belegen mit Nachdruck die Bedeutung der Ausrottung von Polio in den verbleibenden Endemie-Ländern durch systematische Anstrengungen. Es ist absolut notwendig, Polio weltweit auszulöschen, um ein erneutes Einschleppen in andere Länder zu verhindern.
Erfreulicherweise finden diese Maßnahmen auch politische Unterstützung. Bundesminister Niebel forderte auf dem Weltwirtschaftsgipfel in Davos kürzlich:“ Das Beispiel Polio zeigt, wie erfolgreich der Kampf gegen Krankheiten sein kann, wenn er weltweit aufgenommen wird: Polio ist beinahe ausgerottet – gerade jetzt aber, auf der Zielgeraden, darf die internationale Gemeinschaft in ihren Anstrengungen nicht nachlassen. Nur dann können wir die Polio endgültig besiegen“.
Dr. Sabine Diedrich ist die Leiterin des Nationalen Referenzzentrums für Poliomyelitis und Enteroviren (NRZ PE).
Professor Reinhard Burger ist der Präsident des Robert-Koch-Instituts (RKI).
Titelbild: UNICEF (CC BY 2.0).
-

Anonymität in Zeiten kommerzieller DNA-Analysen
Ich kann mit hoher Wahrscheinlichkeit erraten, wie dein Ur-Ur-Ur Großvater – väterlicherseits – mit Nachnamen hieß: Genauso wie du. Was trivial klingt hat kulturelle Hintergründe. Traditionell nehmen Ehepaare bei der Hochzeit den Nachnamen des Bräutigams an, und die Kinder heißen dann ebenso. Nicht nur der Nachname wird so über Generationen weitergegeben, auch das Y-Chromosom männlicher Nachkommen stammt immer vom Vater, und der hat es von dessen Vater, und so weiter.
Genealogie heißt die Erforschung der Abstammungsverhältnisse. Es ist eine Hilfswissenschaft, die wohl vor allem von Großvätern mit viel Zeit ausgeübt wird, und in den USA überaus populär ist. Seit ein paar Jahren wird die Genealogie durch moderne DNA Sequenziermethoden unterstützt. In großen, öffentlichen Datenbanken wie Ysearch und SMGF werden Informationen zu kurzen, sich wiederholenden aber individuell sehr unterschiedlichen DNA Sequenzen des Y-Chromosoms gespeichert, sowie die dazugehörigen Nachnamen. Das hilft den Garagenahnenforschern, etwas über die eigenen Wurzeln heraus zu finden. Man lässt kommerzielle Unternehmen die eigenen sogenannten Short Tandem Repeat (STR) Regionen sequenzieren, und vergleicht die Ergebnisse über eine einfach Eingabemaske dann mit den Einträgen in den Sequenzdatenbanken.Den Nachnamen aus Sequenzdaten bestimmen
DNA Sequenziermethoden werden nicht nur zur privaten Ahnenforschung genutzt. Es gibt große, wissenschaftliche Studien mit dutzenden bis tausenden Teilnehmern, bei denen die DNA der Probanden möglichst komplett sequnenziert wird, beispielsweise um einen Eindruck von der generellen Variabilität menschlicher DNA zu bekommen, oder um bestimmte phänotypische Eigenschaften Unterschieden in der DNA zu zu ordnen. Die Teilnehmer dieser Studien werden in den allermeisten Fällen anonymisiert, so dass durch die Analyse der DNA Sequenzen kein Rückschluss auf die Identität des Teilnehmers möglich ist – oder möglich sein sollte.
Letzte Woche wurde ein Paper in Science publiziert (Gymrek et al.), in dem berechnet wurde, wie hoch das Risiko ist, den Namen eines Probanden zu identifizieren – nur durch die Nutzung öffentlich zugänglicher Datenbanken und durch Internetsuchen. Die Autoren zeigen an einem Testset, dass ihr Algorithmus optimal eingestellt 12% der Namen korrekt identifiziert (5% falsch positiv, 83% unbekannt). In Kombination mit relativ unspezifischen Informationen wie Geburtsjahr und bewohntem US-Bundesstaat war es den Autoren möglich, die Zahl der möglichen Spender einer DNA Probe auf durchschnittlich ein Dutzend Personen einzuschränken.
Die Ergebnisse der Gruppe aus israelischen und US-amerikanischen Forschern sind nicht erschreckend, sie zeigen aber, das die Anonymität von Teilnehmern an großen DNA-Sequenzierstudien unter Umständen nicht gewahrt bleibt, vor allem wenn zusätzliche persönliche Informationen verfügbar sind, auch wenn diese relativ allgemein sind, wie Alter und Nationalität.Weniger ist mehr: Datenschutz und kommerzielle DNA-Analysen
Es gibt noch eine dritte Gruppe Menschen, die Teile ihrer DNA sequenzieren lassen. Während das Ziel der Ahnenforscher ist, über STRs die eigene Abstammung zu rekonstruieren, sind ein Großteil der privaten Kunden von Sequenzierunternehmen auf etwas ganz anderes aus: Sie interessieren sich für die Wahrscheinlichkeit in Zukunft an bestimmten Krankheiten zu leiden. Dazu werden sogenannte SNPs analysiert, also ebenfalls kurze DNA Sequenzen, die mit dem Auftreten von Krankheiten assoziiert sind. Menschen, die sich durch die Sequenzierung von SNPs über Krankheitsrisiken informieren haben oft gute Gründe, ihre Anonymität zu wahren.
Wie hoch ist also das Risiko, dass durch die Analyse dieser SNP-Daten Rückschlüsse auf die Person möglich sind? Dazu habe ich Bastian Greshake befragt, Gründer von openSNP, einer Plattform auf der die Ergebnisse solcher SNP-Analysen publiziert, analysiert und diskutiert werben können.
WeiterGen: Bastian, ist die Anonymität der Benutzern von openSNP nach der Publikation des Gymrek-Papers noch gewährleistet?
Bastian Greshake: Ich vermute das es aktuell nicht so einfach wäre die Benutzer von openSNP mit Nachnamen anreden zu können (also wenn sie ihn nicht angegeben haben). Komplett ausschliessen kann man das natürlich nicht. In dem Paper dort nutzen sie die Haplotypen von bis zu 60 Y-chromosomalen Short Tandem Repeats, darin steckt, meiner Ansicht nach, um einiges mehr an Ancestry-Information, als man über die SNPs die 23andMe auf dem Y-Chromosom testet bekommt (openSNP nutzt hauptsächlich SNP-Daten von 23andMe-Analysen, WG).
WG: Was wäre nötig, um die Anonymität der openSNP Benutzer zu gefährden?
BG: Falls entsprechende Referenzdaten zur Verfügung stünden, könnte man theoretisch von den SNPs aus die Y-STRs imputen, also aus den SNPs die Y-STRs vorhersagen und dann die in der Publikation benutzten Methoden verwenden um die Identität zu ermitteln. Alternativ könnte man direkt Namensdatenbanken verwenden, die SNP-Daten anstelle von STRs verwenden. Diese sind aber derzeit noch nicht weit verbreitet, oder zumindest nicht öffentlich.
WG: Das Risiko, dass aktuell aus SNP-Daten Rückschlüsse auf Einzelpersonen gezogen werden können ist also sehr gering Was können openSNP Kunden dennoch selbst tun, um ihre Anonymität zu wahren?
BG: Ganz generell gilt: Je weniger Metadaten über die Person mit den SNP-Daten verknüpft sind desto geringer die Wahrscheinlichkeit einer Zuordnung. Um die eigene Anonymität zu wahren, sollte man also beispielsweise darauf verzichten sein Alter und seinen Wohnort anzugeben. Angaben dazu sind auf openSNP freiwillig.
Weitere Artikel im Blog zum Thema:-
Es wird immer billiger: Kommerzielle DNA Sequenzierung zur Vorhersage von Krankheiten
-
Können Gene Leben retten? Debatte über personalisierte DNA-Analyse
-
Ein Haufen Daten und doch kein Müll: Das ENCODE-Projekt
-
1000 Genome sequenziert und immer noch nichts passiert
-
Die Risiken des Wissens – Wie sind meine DNA-Daten geschützt?
-
Krankheiten vorhersagen: 23andMe, deCODEme und Navigenics
Titelbild Rosie Cotton (CC BY-NC-SA 2.0).
 Gymrek, M., McGuire, A., Golan, D., Halperin, E., & Erlich, Y. (2013). Identifying Personal Genomes by Surname Inference Science, 339 (6117), 321-324 DOI: 10.1126/science.1229566
Gymrek, M., McGuire, A., Golan, D., Halperin, E., & Erlich, Y. (2013). Identifying Personal Genomes by Surname Inference Science, 339 (6117), 321-324 DOI: 10.1126/science.1229566 -
-
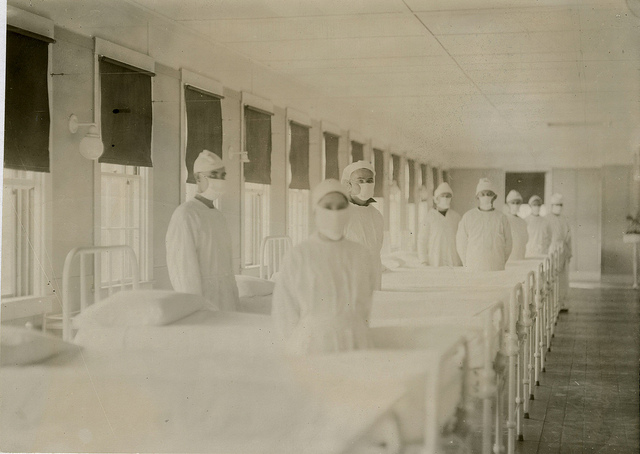
Verrückte Amateurbiologen züchten Killerviren in Garagenlaboren
Am Sonntag erschien in der FAZ online ein aus dem amerikanischen von Volker Stollorz übersetzter Artikel zum Ende des Forschungsmoratoriums an einem Influenzavirus vom Typ A/H5N1. Ein Risikokommunikator nimmt darin Stellung zur Gefahr der Freisetzung des Erregers, zum Beispiel durch einen Unfall oder durch einen dem Wahnsinn anheim gefallenen Wissenschaftler, der an dem Virus forscht.
Der Risikokommunikator bemängelt, dass keine „echte“ Diskussion in der Zeit des Moratoriums (immerhin ein Jahr) über die Gefährlichkeit des Erregers und die Risiken der Forschung daran geführt wurde. Er führt an, dass das Moratorium die Forschung an gefährlichen Viren nicht sicherer gemacht habe. Peter Sandman, der Autor des Artikels, hat in seinem sicher gut gemeinten Artikel leider weitgehend das Thema verfehlt. Zumindest hat er für eine Biosicherheitedebatte den falschen Aufhänger gewählt.
Das eigentlich wichtige Ergebnis der Studie war nicht, dass im Labor ein potentiell gefährliches Grippevirus entstanden ist, welches durch Tröpfcheninfektion zwischen Säugetieren übertragen werden kann. Das wichtige Ergebnis war, wie einfach das Virus gefährlich geworden ist: Fünf simple Punktmutationen haben gereicht, dass ein in Vögeln endemisches Virus plötzlich von Frettchen zu Frettchen übertragen werden kann. Punktmutationen kommen in der freien Wildbahn deutlich häufiger vor, verglichen etwa mit dem Entstehen neuer Virenstämme durch Rekombination.
Das heißt: Die Wahrscheinlichkeit ist groß, dass einige solcher Stämme bereits in ihren Wirten das draußen existieren und nur wenig (Zeit) zur Übertragung auf den Menschen fehlt. Wozu da aber eine freiwillige Forschungspause von einem Jahr gut sein soll, will mit nicht einleuchten. Lars Fischer hat dieses Argument in seinem Blog aufgerollt und kommt ebenfalls zu dem Ergebnis, dass das Moratorium in diesem Fall kontraproduktiv war.
Doch zurück zum Artikel in der FAZ. Der Autor hat in einem Punkt Recht. Das Moratorium wurde nicht für eine ausführliche Debatte über Biosicherheit genutzt, insbesondere über das sogenannte Dual Use Dilemma. Übersetzt auf die Forschung mit pathogenen Erregern heißt das: In wie weit sollen wissenschaftliche Ergebnisse der Allgemeinheit zugänglich gemacht werden, wenn diese in den falschen Händen dazu benutzt werden können Biowaffen herzustellen?
Nun, eben jene Debatte fand vor gut einem Jahr schon bei mir im Blog statt. Ich habe damals meine Leser um ihre Meinung gebeten: Was soll mit den H5N1 Daten geschehen? Etwa die Hälfte der 339 abgegeben Stimmen entfiel auf die Option „Publikation mit modifiziertem Ergebnis- und Methodenteil“ – also so, dass die Ergebnisse zwar öffentlich sind, aber gewisse Hürden existieren, die es nicht jedem Labor ermöglichen die Experimente zu wiederholen. Exakt ein Drittel der Teilnehmer der Umfrage stimmten für die Forschungsfreiheit und die Publikation der Manuskripte ohne Einschränkung. Nur 15% waren der Meinung, man sollte von der Publikation gänzlich absehen.
Weltkarte der Hochsicherheitslabore (S4). Labore in Betrieb sind rot. Eine zoombare Karte gibt es hier.Ein weiterer Aspekt, den Sandman in seinem Artikel in der FAZ behandelt, betrifft die generelle Sicherheit der Forschung an gefährlichen Erregern. Leider taugt die aktuelle Diskussion um die Influenzaviren dafür nur bedingt, obwohl es mehrere Medien, die von Killerviren oder Supervirern sprechen, gerne bedrohlich hätten. Die Viren sind einfach nicht gefährlich genug. Nicht einmal die Frettchen, die sich im Labor mit den mutierten H1N1 Viren infizierten, starben.
Gefährliche Erreger gibt es dennoch wahrlich genug. Ebola, Lassa und Marburg-Viren zum Beispiel verursachen hemorrhagisches Fieber. Eine Ansteckung verläuft fast immer tödlich. Weltweit gibt es nur rund 30 Labore (vier davon in Deutschland) an denen mit solchen Organismen geforscht werden darf. Solche Labore sind komplett abgeschirmt oder gleich in eigenen Gebäuden angesiedelt. Diese Labore der Sicherheitsstufe S4 haben ihr eigenes, geschlossenes Ventilationssystem und drinnen herrscht Unterdruck, so das selbst bei einem Leck nichts entweichen kann. Wissenschaftler, die in solchen Räumen arbeiten, müssen mehrere Schleusen passieren, bevor sie die Räumlichkeiten betreten oder verlassen können und tragen einen Überdruckanzug im Labor. Hier im Video gibt’s das ganze in Bildern (ab min. 6:45).
Man weiß also mit gefährlichen Erregern umzugehen und Laborunfällen ist vorgebeugt. Bleibt einzig die Gefahr des Diebstahls pathogener Keime durch Laborangestellte. Denn die von Sandman beschworenen Amateurbiologen, die in Garagenlaboren Killerviren züchten, die gibt es nur im Film.
Bild oben: Die Influenzaabteilung in einem Marinehospital in den USA 1918 (NavyMedicine, CC BY 2.0)
-

Forschungsfinanzierung durch private Spenden
Kreativität bei der Bewerbung um Forschungsmittel beschränkt sich häufig auf mehr oder weniger weit her geholte Assoziationen des eigenen Forschungsvorhabens mit Krankheiten. Die Assoziationen werden mit Zahlen und Fakten untermauert, in Anträge geschrieben und an Forschungsorganisationen wie die DFG geschickt. Seit vergangener Woche wissen wir, dass die Wege, Forschungsgelder einzutreiben nun um eine Option erweitert wurde. Sciencestarter ist online. Das ist eine deutsche Crowdfundingplattform für Projekte aus der Wissenschaft.
Ob Sciencestarter den gewünschten Erfolg bei der Finanzierung wissenschaftlicher Forschungsprojekte haben wird, ist fraglich, fällt doch der Hauptanreiz von Crowdfunding bei Sciencestarter fast gänzlich weg: Es gibt keine adäquate Gegenleistung für das investierte Kapital. Letztendlich kommen also Investitionen in Projekte Spenden gleich. Die Untersuchung eines Refernzprojekts ergab dann auch, dass ein guter Teil des crowd-gefundeten Geldes von Personen aus der näheren Umgebung der Forscher stammen, also von der Familie, von Verwandten und von Freunden aus sozialen Netzwerken – und nicht etwa von der wissenschaftsinteressierten Allgemeinheit.
Aber die Finanzierung von Forschung aus privaten Quellen muss ja nicht bei einer Crowdfundingplattform aufhören. Gestern erschien ein Artikel in der spanischen Tageszeitung „La Vanguardia„mit dem Titel: „Mikrospender der Wissenschaft“ (Teil 1, Teil 2). Der Artikel führt aus, wie Verwandte und Hinterbliebene von Patienten für die Forschung spenden, zum Teil aus vorhandenem Kapital, zum Teil die Erlöse aus dem Verkauf selbstgemachter Artikel. Große Kliniken in Katalonien schöpfen demnach bis zu 30% ihres Forschungsbudgets aus solchen Quellen. Auch hier kommt das Geld aus der persönlichen Umgebung Betroffener, auch hier spielen soziale Netzwerke beim Fundraising eine Rolle.
Jährliches Spendenaufkommen für Forschungsprojekte beim „Marató de TV3“ in Katalonien. Daten von Wikipedia.
In Katalonien gibt es noch andere Wege, wie Spenden für die Wissenschaft gesammelt werden. Einmal im Jahr, am letzten Sonntag vor Weihnachten, findet auf TV3, einem lokalen Fernsehkanal, ein Spendenmarathon statt, der sogenannte „Marató de TV3„. Jedes Jahr hat wird für eine bestimmte Art von Krankheit gesammelt (dieses Jahr, am 16.12. ist es Krebs), und es kommen innerhalb eines Fernsehnachmittags und -Abends Millionenbeträge zusammen, die direkt den forschenden Laboren zu Gute kommen (siehe eingebundene Grafik oben).
Meine Wahlheimat ist selbstverständlich nicht der einzige Ort der Welt, an dem private Spenden für die Wissenschaft gesammelt werden. Während meines letztjährigen Besuchs des Weizmann-Institut in Israel ist mir aufgefallen, dass fast alle Gebäude die Namen großzügiger Spender tragen. Die Instituts-Website weiß mehr: Das Weizmann hat ein ausgewiesenes Spendenprogramm, innerhalb dessen von Kleinspenden über Einzelstipendien, Spenden für Material und Equipment bis zu kompletten Lehrstühlen und eben auch Institutsgebäude gestiftet, gespendet und verschenkt werden können.
Verteilung des Spendenvolumens in Deutschland 2001 anhand einer repräsentativen Umfrage des Deutschen Spendenrats (Quelle)
Das Spendenaufkommen in Deutschland ist laut Wikipedia international unterdurchschnittlich. Im Schnitt sind es jährlich zwischen drei und fünf Milliarden Euro. Rund drei Viertel dieses Geldes geht in die humanitäre Hilfe. Weitere relevante Posten sind die Kultur- und Denkmalpflege, der Tierschutz und der Umweltschutz. Von Wissenschaft und Forschung weit und breit nichts zu sehen.
Wenn ich als Privatspender für die Wissenschaft spenden wollte, wüsste ich auch gar nicht an wen. Aber jetzt gibt es ja Sciencestarter.Verwandte Artikel im Blog:
-
500 Euro für eingeschweißte Pferdescheiße
-
Petition zur Sicherung des EU Foschungsbudgets unterschreiben
-
Die internationale Finanzkrise als Chance für den Wissenschaftsstandort Deutschland
-
Der Süden führt. Forschungsinvetitionen in Deutschland
-
Wissenschaftsstandort Deutschland. 26 Milliarden für die Grundlagenforschung
-
-
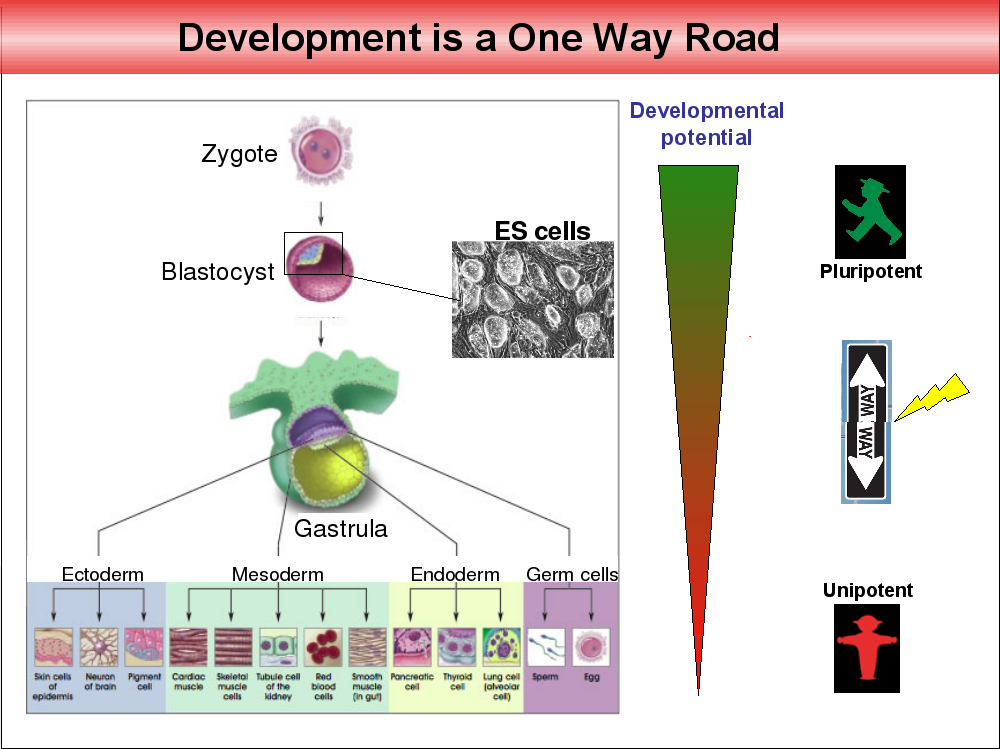
Die biologische Uhr zurück drehen. Nobelpreis für die Reprogrammierung von Stammzellen
Es gibt über 200 verschiedene Zelletypen im menschlichen Körper. Hautzellen, rote Blutkörperchen, Nervenzellen, Darmepithelzellen, Herzmuskelzellen, und so weiter. Vier Tage nach Befruchtung fangen die Zelltypen an, sich zu differenzieren, nach zwei Wochen sind Ectoderm, Mesoderm, Endoderm und die Geschlechtszellen angelegt, zwei Monate nach der Befruchtung sind die unterschiedlichen Zelltypen größtenteils ausdifferenziert. Lange wurde angenommen, dass dieser zelluläre Entwicklungsprozess einer Einbahnstraße gleicht, der mit pluripotenten Zellen beginnt, Zellen also, die das Potential haben, sich in viele verschiedene Zelltypen zu differenzieren, und in unipotenten, ausdifferenzierten Zellen mit einer definierten Funktion endet.
Erst vor ein paar Jahren gelang es Shin’ya Yamanaka diesen Prozess der Ausdifferenzierung umzukehren. Er konnte Bindegewebszellen von Mäusen in Labor durch Gabe bestimmter Transkriptionsfaktoren (sie heißen Oct3/4, Sox2, Klf4 und c-Myc) in einen Zustand überführen, in dem die Zellen sich erstaunlicherweise wieder in andere Zelltypen ausdifferenzieren liesen. Die biologische Uhr der Differenzierung wurde sozusagen zurück gedreht. Die so geschaffenen Zellen wurden induzierte pluripotente Stammzellen, oder iPS-Zellen getauft.
Diese erstaunlichen Ergebnisse wurden inzwischen von vielen Laboren bestätigt und erweitert. Es werden immer weitere Möglichkeiten entdeckt, differenzierte Zelltypen in deren Vorläuferzellen zurück zu verwandeln oder bestimmte Zelltypen ineinander umzuwandeln. Die Methoden werden verfeinert und Stück für Stück werden so die molekularen Grundlagen der Zellentwicklung aufgedeckt.
Hoffnungen für den therapeutischen Nutzen von iPS-Zellen werden ebenfalls an die induzierten pluripotenten Stammzellen geknüpft. Beispielsweise wäre es denkbar, körpereigene Haut für Brandopfer oder neue Herzmuskelzellen für Infarktpatienten aus iPS Zellen herzustellen. Nicht zuletzt wurde die Entdeckung der Reprogrammierung von Gegnern der Forschung an embryonalen Stammzellen als ein Argument ins Feld geführt, dass man auf richtige Stammzellen nun ja in der Forschung verzichten könne. Experten sind anderer Meinung.
Der diesjährige Nobelpreis für Physiologie und Medizin wurde zurecht an Shin’ya Yamanaka verliehen. Seine Forschung steht für einen wissenschaftlichen Paradigmenwechsel mit noch unabsehbaren Konsequenzen, inklusive therapeutischer Anwendungen. Zum zweiten Preisträger dieses Jahr, John Gurdon, mehr heute Nachmittag.
Die Abbildung oben stammt aus einem Vortrag von Konrad Hochedlinger. -
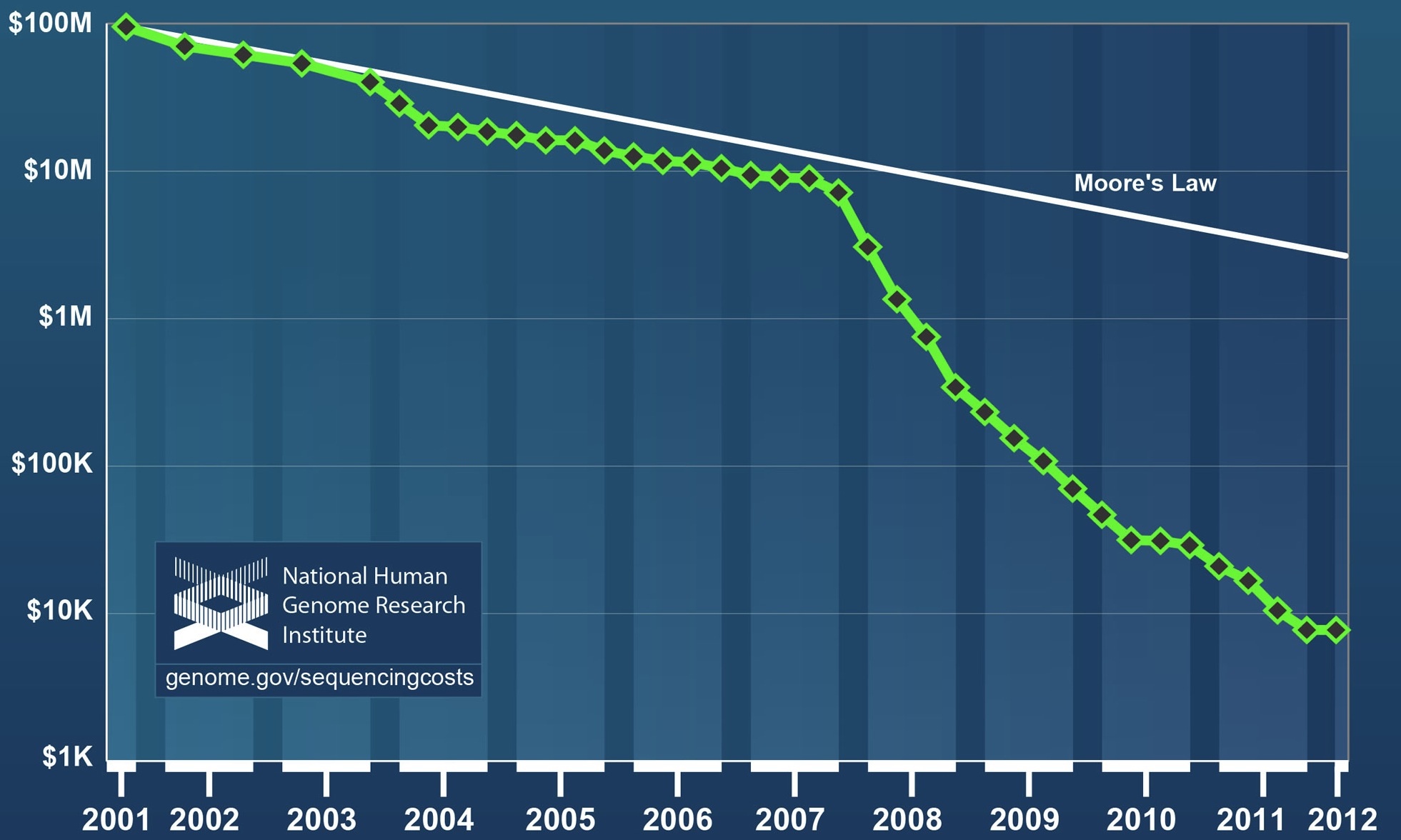
Es wird immer billiger: Kommerzielle DNA Sequenzierung zur Vorhersage von Krankheiten
Vor zwei Wochen nahm ich an einer Diskussionsrunde zu personalisierten genetischen Tests Teil. Firmen wie 23andMe bieten für ein paar Hundert Dollar an, die eigene DNA zu analysieren und dann Rückschlüsse auf Krankheitsrisiken, aber auch auf die persönliche Abstammung zu ziehen. Ich habe hier im Blog von der Diskussionsrunde (live) berichtet.
Im Zuge meiner Vorbereitungen habe ich aktuelle Angebote für diese personalisierten genetischen Tests recherchiert. 23andMe testet derzeit für 300 Dollar. Das sind 100 Dollar weniger als vor knapp drei Jahren. Günstiger ist natürlich gut, und auch die Zahl der ausgewerteten Merkmale ist von rund 120 vor drei Jahren auf derzeit 243 gestiegen. Insgesamt werden dafür von 23andMe Daten von rund einer Million SNPa analysiert. Die Technik, die 23andMe verwendet nennt sich Genotypisierung. Dabei werden Mutationen in kurzen DNA-Abschnitten (single nucleotide polymorphisms, SNPs), Wahrscheinlichkeiten für bestimmte Krankheiten zugeordnet. Die Wahrscheinlichkeiten wurden (und werden weiter) in sogenannten genomweiten Assoziationstudien (GWAS) bestimmt.
Die Genotypisierung ist nicht die einzige Möglichkeit, Daten zu Krankheitswahrscheinlichkeiten durch die Analyse der DNA zu gewinnen. Die Alternative ist die DNA Sequenzierung. Und hier sind die Kosten in den letzten Jahren dramatisch gefallen. Die Grafik des National Human Genome Research Institutes zeigt detailliert, wie sich die Kosten für die DNA Sequenzierung seit 2001 entwickelt haben. Der Preisverfall übeflügelt das Moorsche Gesetz weit und besonders auffällig ist der Preisknick nach 2007 (siehe Abbildung oben). Dieser hängt mit einer technischen Neuerung zusammen: Das sogenannte Next Generation Sequencing hat vor ein paar Jahren das traditionelle Sanger-Sequenzing in den großen Sequenzierzentren abgelöst. Das Next Generation Sequencing unterschiedet sich vor allem durch eine massive Parallelisierung der Sequenziervorgänge, hier ein Übersichtsartikel über Sequenziertechniken.
Weltweite Verteilung der Next Generation Sequenziermaschinen, Stand 09/2012. Quelle http://omicsmaps.comInteressant ist auch die weltweite Verteilung der Next-Generation Sequenziermaschinen, sie bildet annährend die Verteilung der Wissenschaftsausgaben ab. Aktuell sind 2035 dieser Sequenziergeräte im Einsatz. 922 davon stehen in Nordamerika, 604 in Europa und 377 in Asien. Das weltweit größte Sequenzierzentrum ist das BGI in China mit 166 Maschinen. In Deutschland stehen 142. Trotz dieser Explosion an Sequenzierpower kostet aktuell die Sequenzierung eines kompletten menschlichen Genoms noch deutlich zu viel, um kommerziell mit SNP-Genotyping konkurrieren zu können. Das sogenannte Exomesequening, bei nicht das ganze Genom sequenziert wird, sondern nur die Teile, die tatsächlich für Proteine kodieren, ist jedoch ein günstigerer Zwischenschritt, der mittelfristig die Genotypisierung wenn nicht ganz ablösen, doch zumindest ergänzen wird. 23andMe muss derzeit dennoch keine Angst vor Konkurrenz haben. Das Unternehmen bietet (für bestehende Kunden) die Sequenzierung des Exoms mit 80-facher Coverage bereits für 999 Dollar an.
Trotz aller Preisstürze: Deutlich günstiger und häufig zuverlässiger als genetische Tests zur Vorhersage von Krankheiten sind Blutdruck messen, ein Blick auf den Bauchumfang und auf persönliche Laster.
Quelle Bild oben: Wetterstrand KA. DNA Sequencing Costs: Data from the NHGRI Large-Scale Genome Sequencing Program www.genome.gov/sequencingcosts.
-

Wann verlassen mit Genmais gefütterte Ratten das sinkende Schiff?
Im Englischen gibt es einen schönen Ausdruck, für den ich kein deutsches Äquivalent kenne: Preaching to the choir. Vielleicht trifft es offene Türen einrennen am besten. Oder Eulen nach Athen tragen? Jedenfalls habe ich das Gefühl, wenn ich hier über das Séralini-Paper (hier das .pdf) von letzter Woche schreibe, sowieso schon jeder weiß worum es geht und wo die Probleme bei der Studie liegen (hier noch ein paar Expertenmeinungen): Schlechtes Studiendesign, eine seltsame oder fehlende statistische Auswertung und das Vorenthalten von Daten. Ich würde mich nicht wundern, wenn Séralini das Paper demnächst zurück ziehen müsste. Aber der Medien-Coup, das Bewerben seines Buches, ist ihm mit Sicherheit gelungen. Gentechnisch veränderte Pflanzen waren ja auch hier im Blog schon manchmal das Thema und dieser Artikel mit Gründen für Grüne Gentechnik wird gerade wieder häufiger aufgerufen, wenn ich den Statistiken on Google Analytics glauben kann.
Insgesamt hatte ich trotzdem den Eindruck, dass die Presse dieses Mal relativ schnell den Braten gerochen hat, und daher weniger sensationslüstern über die Fütterungsstudie an Ratten berichtet hat. Die Achse des Guten hat dennoch ein paar Genmais is böse und macht Krebs-Links gesammelt. Eine Übersichtsseite mit Links zu Artikeln hat auch Marcus Anhäuser vom Mediendoktor angelegt. Ein aktueller Artikel in Slate vergleicht die Gegner genetisch veränderter Organismen mit Klimaskeptikern. Nur das die einen eher rechts und die anderen eher links im politischen Spektrum anzusiedeln seien. Eine interessante Parallele, wie ich finde.
Wer hat noch interessante Links gefunden? Oder unkritische Artikel?
Und – was völlig anderes – vor drei Wochen sind wir ja mit ScienceBlogs auf WordPress umgestiegen. Was nervt? Was fehlt? Was ist besser?
Schönes Wochenende.
-
Radikalisierte Terrorkommandos – Was verursacht Brustkrebs?
Spätestes seit Cornelius‘ Artikel vom Vormonat wissen wir, dass Krebs „der Feind im eigenen Körper [ist], der aus einer irgendwann entarteten […] Zelle hervorgegangen ist, die sich Schritt für Schritt zu einem mit allen Wassern gewaschenen Terrorkommando […] verwandelt hat„.
Es sind Mutationen, die für Krebs verantwortlich sind. Mutationen, die dazu führen, dass sich Zellen unkontrolliert teilen. In Nature ist jetzt eine Studie des Cancer Genome Atlas Consortiums erschienen, in der systematisch die molekularen Grundlagen von Brustkrebs analysiert und kartiert wurden. Oder um in Cornelius‘ dichterischem Duktus zu bleiben: In der Studie wird untersucht, welche Terrorkommandos im Busen zum Einsatz kommen und was diese radikalisiert.
Brustkrebs ist der bei Frauen am häufigsten diagnostizierte bösartige Tumor. In Deutschland sterben pro Jahr über 17 000 Menschen an den Folgen von Brustkrebs und jedes Jahr werden 72 000 neue Fälle diagnostiziert. Da nur etwa 5% aller Brustkrebserkrankungen erblich bedingt sind, stellt sich die Frage, welche für Brustkrebs verantwortliche Mutationen in welchen Genen im Lauf eines Lebens auftreten. Ein besseres Verständnis der molekularen Grundlagen von Brustkrebs erlaubt einerseits eine bessere Klassifizierung diagnostizierter Tumore und andererseits viel zielgerichtetere Therapien, beispielsweise mit Antikörpern und Hormonantagonisten.
Die Autoren der Studie, ein großes Konsortium hauptsächlich in den USA ansässiger Forschergruppen, haben Tumorzellen und zur Kontrolle Keimbahnzellen von 825 Patienten mit einer ganzen Batterie von molekularen Hochdurchsatzverfahren untersucht. Gesammelt wurden mRNA Expressionsdaten, Informationen zur DNA Methylierung und zu DNA Punktmutationen. Weiter wurde die microRNA sowie das komplette Exom sequenziert und die Expression bestimmter Proteine gemessen. Für 348 Tumore waren schließlich Daten von allen angewandten Techniken vorhanden.
Mammakarzinome können klinisch in drei therapeutische Gruppen eingeteilt werden. ER-positive Tumore sprechen auf Hormonantagonisten an. Brustkrebszellen, in denen mehrere Kopien des HER2 Gens gefunden werden, können mit Antikörpern therapiert werden. Eine dritte Klasse, der diese Marker fehlen, sind auf Chemotherapie angewiesen. Diese Klassifizierung konnte in den letzten Jahren durch Genexpressionsanalyse auf vier Subtypen erweitert werden. Diese vier molekularen Klassen konnten in der jetzt vorliegenden Studie bestätigt und verfeinert werden.
Eines der Schlüsselergebnisse ist, dass viele der neu entdeckten Brustkrebsmutationen spezifisch in den einzelnen, bereits etablierten Subklassen auftraten. Die Integration mit den Ergebnissen der anderen Analysemethoden erlaubt jetzt deutlich detailliertere Einblicke, was auf molekularer Eben in den Brustkrebsklassen passiert. Eine Zusammenfassung bietet diese Tabelle.
Zudem sollten diese Ergebnisse ermöglichen, die Brustkrebsdiagnose durch Einsatz komplementärer Techniken zu verbessern. Eine Maßnahme, die Leben retten kann. Matthias Mann, Direktor am MPI für Biochemie und einer der weltweit führenden Proteomforscher spricht davon, dass derzeit 10% aller diagnostizierten Brustkrebsfälle falsch klassifiziert werden.
Interessanterweise fanden die Autoren der Nature Studie außerdem, dass eine der etablierten Brustkrebsklassen, der sogenannte basal-like-Subtyp, eine ähnliche molekulare Signatur aufweist wie eine Form des Ovarialkarzinoms. Die Autoren folgern, dass beide Krebstypen folglich auf die gleichen therapeutischen Ansätze reagieren sollten.
Die Studie ist online bei Nature frei zugänglich. Das Foto oben ist von tipstimes (CC BY-SA 2.0). Zur Diskussion ob dieses Photo eine sehr ernste Krankheit unnötig sexualisiert hier entlang.
 The Cancer Genome Atlas Network (2012). Comprehensive molecular portraits of human breast tumors Nature DOI: 10.1038/nature11412
The Cancer Genome Atlas Network (2012). Comprehensive molecular portraits of human breast tumors Nature DOI: 10.1038/nature11412 -

Wie Patienten von Open Data profiteren könnten
Das Hauptthema war das gleiche wie seit Jahren in den Blogs. In der Session zu „Open research and education“ auf dem Open Knowledge Festival in Helsinki wurde viel über Open Access diskutiert, also um den kostenlosen Zugang zu wissenschaftlicher Literatur für alle, im Idealfall mit damit verbundenen Rechten, die Daten weiter verwerten zu können.
Selbstverständlich ist Open Access wichtig und die Kritik an dem Geschäftsgebaren der großen, kommerziellen Wissenschaftsverlage angebracht: Es ist nicht ersichtlich, worin genau die Wertschöpfung der Verlage besteht, um die horrenden Abopreise für Wissenschaftsmagazine zu rechtfertigen. Das hier keine wirksamen Marktmechanismen greifen, ist unter anderem an der Breite der Preise für Einzelartikel der Verlagshäuser ersichtlich (Abbildung unten). Wenn sich Teilnehmer einer Konferenz zu Open Date gegenseitig vom Nutzen von Open Access überzeugen wollen, gleicht das allerdings dem Einrennen offener Türen, wie einer der Teilnehmer in einer der Diskussionsrunden kritisch anmerkte.
Artikelkosten und deren assoziierten Publikationslizenzen. Open Access ist nicht teuer. Abbildung von Ross Mounce, CC by 3.0Wie Open Data den Wissenschaftsbetrieb beeinflussen kann, wurde an zwei weiteren Beispielen versucht aufzuzeigen. Ein Labor in Australien führt öffentliche Laborbücher im Wiki-Format. Inititativen wie Figshare fungieren als frei zugängliches Repositorium für wissenschaftliche Daten mit Fokus aus deren visuelle Darstellung, sei es als Abbildung, Animation oder Video. In wie weit diese sicher sehr löblichen Ansätze Spielerei einiger Idealisten bleiben oder tatsächlich beeinflussen, wie wir in Zukunft Wissenschaft betreiben, publizieren und kommunizieren, ist von der Reform der gängigen vertraglichen Rahmenbedingungen hinsichtlich des geistigen Eigentums der Forschungsergebnisse abhängig – und nicht zuletzt von den Geschäftsmodellen und Finanzierungsmöglichkeiten der Open Data Bewegung.
Ich würde mir wünschen, dass bei kommenden Konferenzen zur Rolle von Open Data in der Wissenschaft noch andere, dringendere Aspekte thematisiert werden: Wie und wo werden wissenschaftliche Datensätze standardisiert gespeichert und frei zugänglich, so dass diese auch langfristig und ohne Qualitätsverlust interpretierbar bleiben? Wie können komplexe Daten visualisiert werden, so dass auch nicht-Experten von wissenschaftlichen Ergebnissen profitieren können?
Ben Goldacre beschreibt in seinem neuem Buch Bad Pharma
wie pharmazeutische Unternehmen Ergebnisse von Patientenstudien unterschlagen, die nicht den gewünschten Effekt zeigen (ein Auszug hier im Guardian). Es wird nur publiziert, was der Marketingstrategie entspricht. Das ist doch ein Thema wie gemacht für Open Data: Open Pharma. Der direkte Nutzen für Patienten (und Ärzte) ist offensichtlich.