09.55
Guten morgen und Willkommen zum Live-Blog von der Diskussionsrunde zu direct-to-consumer genetic testing.
09.57
Das Wetter heute morgen in Barcelona: Frische 18°C, keine Wolke trübt den strahlend blauen Himmel. Folglich bin ich heute – wie alle Fahrer – auf Trockenreifen angereist. Die Veranstaltung findet am
CRG statt.
09.59
Das Live-Blog Plugin scheint soweit zu funktionieren, wahrscheinlich muss die Seite aber manuell neu geladen werden, um die Updates zu sehen.
10.05
Eine weitere gute Nachricht: Die Speakers trudeln auch ein, der Beamer funktioniert, und es sieht so aus, als könne es demnächst losgehen.
10.13
Nur einer der hier anwesenden hat bislang seine SNPs sequenzieren lassen. Das ist überraschend. Aber es gibt einige, die es sich – wie ich auch – zumindest überlegen.
10.21
Kelly Rabionet gibt jetzt erst mal eine sehr grundsätzliche Einleitung. Zellen – DNA drin – 20 000 Gene – ATCG basen – Sequenzen – Vererbung – Mutationen – Krankheiten. Das ist etwas zu basic für die hier anwesenden und wohl auch für die hier mitlesenden.
10.27
Rabionet erinnert, wie die Tests funktionieren. Bislang wird nicht die ganze DNA sequenziert, sondern nur bestimmte Stellen untersucht, von denen man weiß, dass Nukleotidaustausche mit gewissen Phänotypen korrelieren.
10.36
Rabionet zeigt an graphischen Beispielen, wie die Sequenzierungsdaten grapisch aufbereitet werden. Wer sich das ansehen will, findet die gleichen Slides auf der 23andMe und der deCodeMe Website. Es wird jedenfalls schon eine Herausforderung klar: Wie stellt man die gewonnenen Daten so dar, dass der Verbraucher sie versteht?
10.40
Ein weiteres Problem ist die Einordnung der detektierten Varianzen bezüglich tatsächlicher Krankheiten. Das System basiert auf wissenschaftlichen Publikationen, die diese Phänotypen in Relation zu den Mutationen beschreiben. Jedoch wird da häufig nur ein gewisser Marker oder ein Teil eines betroffenen Gens angeschaut, so dass das Gesamtrisiko einer Krankheit sich durchaus von der Summe der Einzelrisiken – die sicher richtig beschrieben sind – unterscheiden kann.
10.43
Eine Anmerkung aus dem Publikum: Die Bewertung der Risiken können sich selbstverständlich im Lauf der Zeit ändern, beispielsweise wenn neue Studien publiziert werden. Außerdem ist die Konfidenz einer SNP-Phänotyp-Korrelation wohl ausschließlich auf der Anzahl der Patienten in der jeweiligen Kohortstudie basiert.
10.46
Jetzt komme Lluis Armengol. Er war Doktrand und Postoc hier, und ist jetzt CEO von qGenomics. Die Firma biete hauptsächlich Pränataluntersuchungen an. Sie nutzen DNA-Microarrays.
10.51
Er gibt einen Überblick über direct-to-consumer genetic testing Firmen:
- 50-60 Firmen, heterogene Größe
- 5 Milliarden Markt für klinische Diagnostics 2010, steigende Tendenz
- Die meisten sind in den USA und UK, dann Europa
- zwischen 90 und 1110 Dollars (hängt auch von der Zahl der getesteten Marker ab)
- Drei große Gebiete: Gesundheit, Abstammung und Life-Style
10.52
Hier ist Armengol vor einem Slide mit Firmennamen, die diese Services anbieten:
10.59
Unterschiedliche Businessmodelle: Forschungsintensiv (Beispiel decode) vs Serviceorientiert (Beispiel 23andMe) vs. Shops für spezifische Tests (Beispiel Genelex).
Das Geschäftsmodell ist häufig nicht nur der finanzielle Profit durch den Verkauf von Kits, sondern auch die Schaffung von Communities und der (anonymisierten) Auswertung der Kundendaten.
Die größten Players: 23andMe, Decode Genetics, Navigenics, Pathway Genomics, Knome (Illumina) und Myriad Genetics.
11.05
Schwächen der Geschäftsmodelle:
- Möglicherweise wird die Größe des DTC (direct to consumer) Marktes überschätzt (immerhin hat hier im Saal sich bislang nur einer testen lassen – oder gibt dies zumindest zu).
- Und wieder die Frage, wie verlässlich und wie nützlich die Daten tatsächlich für den Verbraucher sind – speziell wenn es keine Möglichkeit gibt, etwas “dagegen zu tun”.
- Anscheinend niedrige Markteintrittshürden (die eigentlichen Analysen werden in Drittlaboren durchgeführt)
- Und hier die Erwähnung von Open Source tools: OpenSNP (Gruß an einen der Co-foundersBastian Greshake
11.13
Armengol ist fertig, Frage aus dem Publikum: Sind die Firmen in den USA FDA approved? Armengol: Nein.
Was sind die wahren Kosten der Firmen pro Kunde? Armengol: Nicht mehr als 200 Dollar. Ich denke, das muss noch weniger sein.
Anmerkung von Pascal Borry: Es gab Ansätze, die Tests umsonst anzubieten, da der wahre Wert in den Daten und in einer großen Anzahl User steckt.
11.15
Jetzt ist eine Pause, wir schlagen uns die Bäuche voll und nachher geht es mit der Diskussion hier weiter. Ich bitte die Aussetzer des Liveblog-Plugins zu entschuldigen, es geht eben noch nicht alles nach Plan.
11.35
Die Exponierte Lage und die Architektur des Gebäudes ist häufig ein Grund für Wissenschaftler, sich hier zu bewerben.
11.37
Es geht weiter mit Pascal Ducournau, Assistenzprofessor für Soziologie in Toulouse. Titel seines Vortrags: Direct to consumer health testing services: what commercial strategies for which socio ethical issues? Da bin ich mal gespannt.
11.38
Noch eine Zahl: 23andMe hatte angeblich 50 000 Kunden in 2010 und 100 000 in 2011.
11.43
Welche Marketingstrategien verfolgen diese Firmen, um Kunden zu gewinnen? Und wie wirken diese soziologisch? Dafür hat Ducournau 42 Firmen untersucht, die DTC testing anbieten. Was sind die Angebote? Wie viele Traits werden untersucht, wie viele Krankheiten direkt? Welche Slogans verwenden die Sites?
11.46
Hauptsächlich Marker für Herz-Kreislaufkrankheiten werden untersucht. Dann Brustkrebs und Pharmakogenomics (also wie reagiert man auf bestimme Medikamente). Weiter werden natürlich Krankheiten untersucht, die relativ eindeutig zu untersuchen sind: Zystische Fibrose, Hematochomatosis.
11.49
Ducournau fügt den Begriff “healthism” ein: Die persönliche Gesundheit ist der Hauptfaktor fürs Wohlfühlen. Erreicht wird das durch die Änderung des eignen Lebensstils, mit oder ohne therapeutische Hilde. Binsenweisheit oder neues soziologisches Konzept? Ich weiss es nicht.
11.57
Ein weiterer Aspekt sei die Personalisierung der Gesundheit. Man nimmt selbst Einfluss auf medizinische Entscheidungen und hängt nicht mehr nur vom Hausarzt ab. Dazu passende Slogans der Firmen:
Your health your right to know, etc.
Das gleiche gälte bezüglich der Forschung: “
Get involved in a new way of doing research”
Der Soziologe erwähnt auch die Rolle des Webs. Durch Blogs, Foren und Chartooms würden persönliche Beziehungen zwischen den Benutzern untereinander und zwischen den Unternehmen und deren Kunden geschaffen. In seinen Worten:
Emergence of new social entities under the effect of biotechnological discoveries and applications called biosocialities.
12.00
Zusammenfassung der Marketingstrategien:
- Die Ausnutzung des Healthisms (wahrscheinlich war Harald Schmidt einer der Erfinder).
- Die Individualisierung der Gesundheit
- Die Bildung digitaler Communities
12.03
Hier die Zusammenfassung seiner Thesen http://tegalsi.hypotheses.org/
Jetzt Pascal Borry. Über Ethik.
12.08
Er fängt mit einer Übersicht der Vor-und Nachteile an und erwähnt das Fehler der professionellen Nachsorge und unbekannte Konsequenzen auf die Gesundheitssysteme. Wichtige Punkte, mal sehen ob noch mehr kommt.
12.16
Welche Maßnahmen können getroffen werden, um das Feld zu kontrollieren? Borry illustriert das mit einem Foto der Daltons – also unkontrollierbare Outlaws. Hier jetzt seine Vorschläge für Regulierung:
- Information der Gesellschaft und der Ärzte was solche Tests leisten können, was nicht und wo die Probleme sind
- Eine Selbstregulation der Industrie, also evtl, ein ethischer Kodex
- Einführung von Standards bezüglich der Werbung
12.24
Ein weitere Punkt, der von Burry angesprochen wird: Die Regulierung der genetischen Tests und die Zertifizierung der Labore (sind die Ergebnisse richtig? Sind die Analysemethoden auf neuestem technischem Stand?). Er führt aus, dass in Frankreich genetische Tests nur zu medizinischen und Forschungszwecken zugelassen sind.
Das ist natürlich schwer umsetzbar. 23andMe zum Beispiel
wirbt direkt damit, auch in andere Länder zu verschicken, unter anderem Frankreich (und auch Deutschland)
12.31
Er führt weitere Länder und deren Regulierung an. Holland, Portugal, die Schweiz. Es gibt auch eine europäische Richtlinie zur Regulierung, die allerdings von vielen Ländern nicht ratifiziert ist (unter anderem Deutschland). Interesanterweise hat Island die Richtlinie unterschrieben, deCodeMe, einer der größten Player im Markt, ist (zum Teil) isländisch und die meisten der sequenzierten Kunden sind Isländer. Mit der Richtlinie wäre dem Unternehmen die rechtliche Geschäftsgrundlage entzogen.
In Frankreich ist man da weiter: Es ist es gesetzlich verboten einen genetischen Test anzufordern. Strafe: 3750 Euro.
12.36
Jetzt Diskussionsrunde. Ich habe auch eine Frage, mal sehen ob ich drankomme: Was passiert wenn Versicherungen Zugang zu den Daten bekommen?
12.38
Erste Frage kommt zum Nutzen: Können solche Tests helfen, Krankheiten zu verhindern? Gibt es die Möglichkeit der Prävention? Ich denke gewissen Änderungen im Lebensstil oder häufigere Vorsorgeuntersuchungen können schon helfen.
12.44
Die erste Frage ist ambivalent beantwortet. Manche denke, man könne durch Tests und das Wissen um eigene Risikofaktoren den Lebensstil ändern und profitieren, andere denken, das würde in die Autonomie des eigenen Lebens eingreifen. Eine Anmerkung war, dass der Nutzen vor allem in Pharmakogenomics liegt. Also: Wie verträgt jemand gewisse Medikamente?
12.59
Meine Frage war, was passiert wenn Versicherungen Zugang zu den Daten bekommen? Oder gar selbst Produkte anbieten, die die Analyse der Daten mit einbeziehen? Die Ethiker reden sich raus: Die Qualität und die Relevanz der Tests ist nicht gut genug. Aktuell. Borry fügt an, dass es andererseits darum geht, etablierte und funktionierende Tests allen zur Vefügung zu stellen, und da seien nicht alle Gesundheitssysteme auf dem selben Level
12.59
Falls jemand der hier mitliest eine spezifische Frage hat: Bitte kommentieren.
13.07
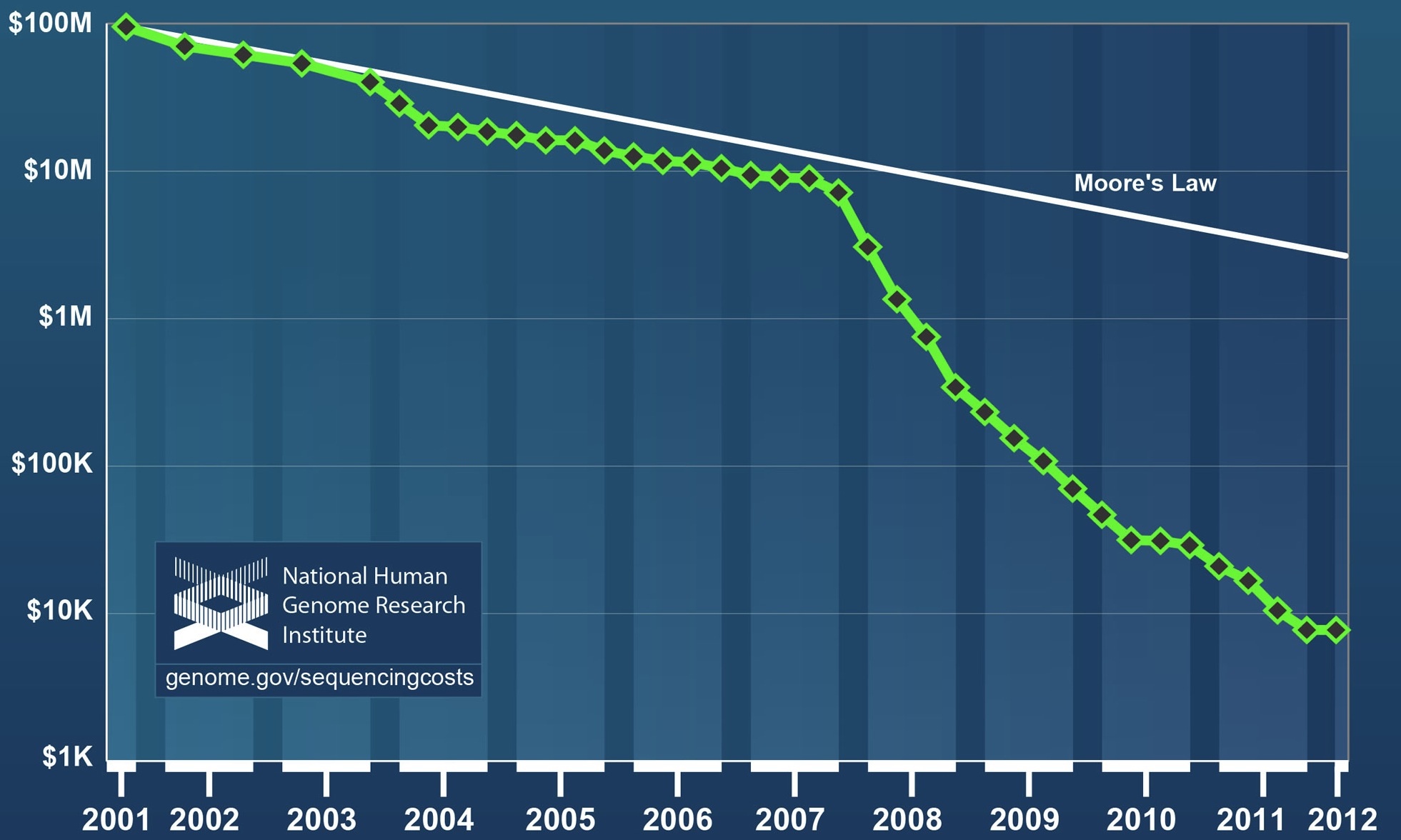
Es gibt zwei Diskussionsstränge: Reicht die Qualität der Tests überhaupt aus zuverlässige Aussagen zu machen? Und wie gehen Gesundheitssysteme mit einer möglichen Datenflut um? Der Grundtenor der Experten scheint zu sein: Technisch sind die aktuellen Genotyping-Tests kein Problem, allerdings veraltet. Exome Sequenzing (also alle mRNA Transkripte nach dem Splicen) ist das nächste. Das Gesamtgenom kommt dann.
Allerdings sind die Wahrscheinlichkeitsvorhersagen für eine Krankheit basierend auf den SNP-Daten problematisch, da viele zusätzliche genetische Faktoren, die eine Rolle spielen könnten nicht berücksichtigt werden, da sie aktuelle entweder nicht sequenziert werden, oder deren Einfluss auf eine gewissen Krankheit unbekannt ist.
13.09
Frage: Wie groß ist der Prozentsatz der Menschen, die sich sequenzieren lassen würden.
Antwort: 5 Promille
Es wurde auch erwähnt, dass sich diese Zahl nicht unbedingt mit dem Wissen ob der Möglichkeiten für die Tests erhöht.
13.17
Gute Frage: Wird es je günstig genug sein, dass alle ihr Genom sequenzieren lassen können und werden die Daten je interpretierbar genug sein, um sinnvolle medizinische Vorhersagen zu treffen?
Kurze Antwort: Günstig ja, sinnvolle Vorhersagen: keine Ahnung.
13.19
Weiterer interessanter Punkt: We hilft Laien die Daten zu interpretieren. Nicht nur SNP Data, sondern zum Beispiel auch pränatale Tests auf Krankheiten bei Schwangeren? Eventuell ergibt sich hier ein neues Berufsbild des “Genetic Counselors”. Ärzte jedenfalls scheinen aktuell mit Diagnose und Interpretation überfordert.
13.20
So, das wars von hier. Die Session ist vorbei und ich gehe Mittagessen. Leider hakt die Einbindung des Liveblogs noch machmal, ich hoffe diese Macken geben sich noch. Danke fürs mitlesen und Kommentare und Fragen gerne weiter unten.