Als Angehöriger einer Krebspatientin ist eines unvermeidbar: Einem werden von vielen Seiten oft gut gemeinte „alternative“ Therapiemöglichkeiten vorgeschlagen.
Kurkuma, Canabisöl, Hemohim, Reservatrol, Methadon, intra-arterielle Chemotherpaie, Bruno Gröning und so weiter. Da ist viel hanebüchenes Zeug dabei, man glaubt es erst, wenn man es selbst hört und liest.
Die gut gemeinten Ratschläge lassen sich in drei Katergorien einordnen. Da sind erstens die Diät-Tips, zweitens die medikamentösen Alternativtherapien und drittens die – nennen wir sie mal – „spirituellen“ Vorschläge zur Heilung.
Vielleicht schreibe ich zu einigen der vorgeschlagenen Alternativen irgendwann noch mal mehr. Ich antworte jedenfalls auf die Vorschläge die uns erreichen damit, dass wir uns entschieden haben, den Weg zu gehen, der erwiesernmaßen lebensverlängernd wirkt: Den der Medizin.
Es ist gar nicht so leicht zu beantworten, um wie viel Jahre die Medizin eigentlich das Leben Krebskranker verlängert. Zum einen müssen unterschiedliche Krebsarten unterschieden werden, zum andern spielen neben dem medizinischen Fortschritt noch Faktoren wie eine bessere Krebsvorsorge und Früherkennung eine Rolle.
Was man jedoch sicher sagen kann ist: Alternativemedizin tötet. Auch zusätzlich zur konventionellen Krebstherapie, also der ganzen oder teilweisen operativen Entfernung vom Tumoren, der Strahlentherapie und der Chemotherapie, wirkt Alternativmedizin nicht. Wer mehr Beispiele braucht sei an das Blog Science Based Medicine verwiesen.
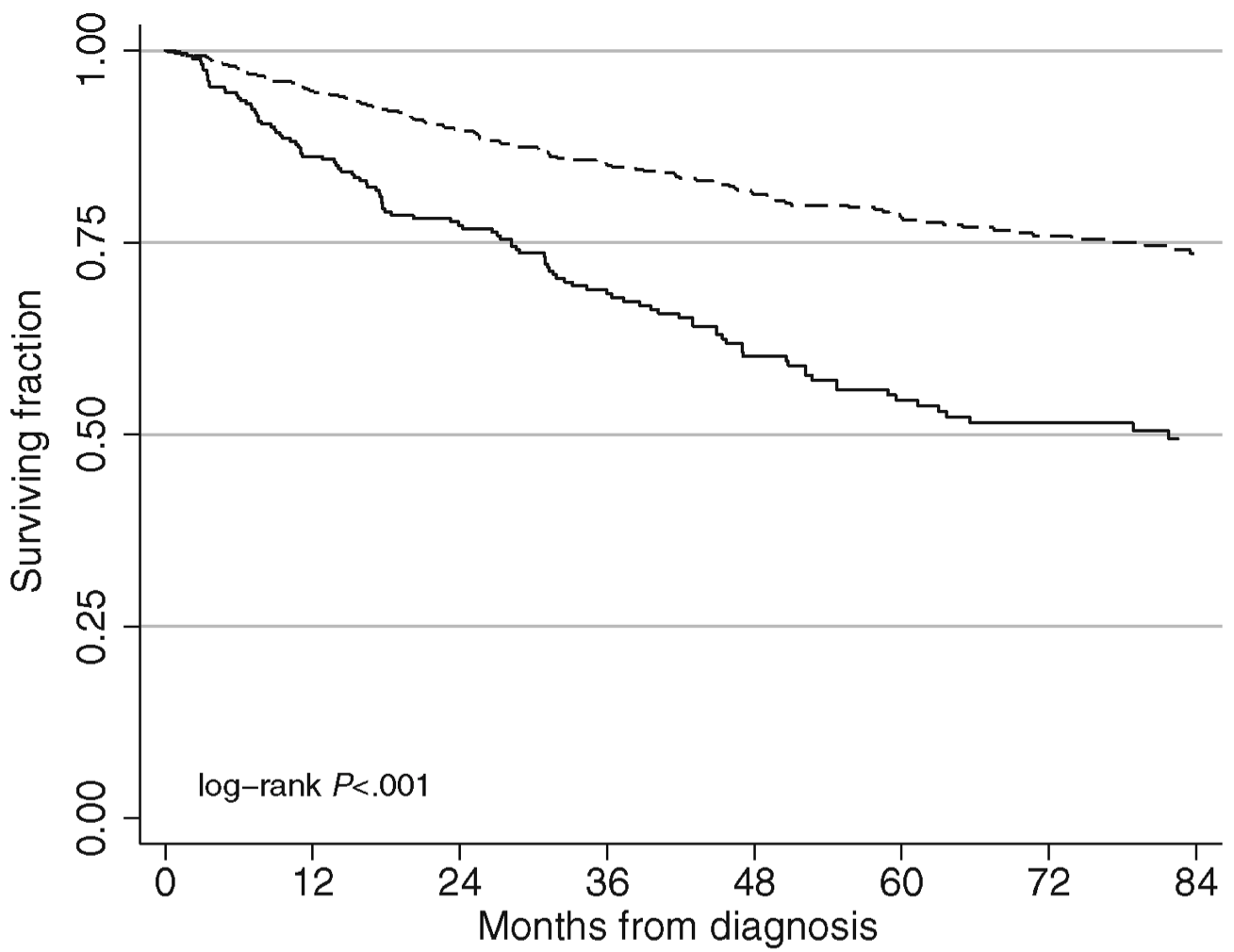
Hier habe ich nur eine Abbildung aus einem Paper vom Januar 2018 eingebunden. Die Abbildung zeigt den Anteil der überlebenden Krebspatienten und Patientinnen über einen Zeitraum von 7 Jahren nach ihrer Diagnose. Die Patienten hatten die häufigsten Krebsarten, also Lungenkrebs, Brustkrebs, Darmkrebs und Prostatakrebs.
Die gestrichelte Linie zeigt die Patienten, die sich auf konventionelle Therapiemethoden verlassen haben. Die durchgezogene Linie zeigt Patienten, die sich statt der konventionellen Therapie auf alternativmedizinische Methoden verlasen habe.
Nach sechs Jahren war die Hälfte der Alternativmedizinpatienten tot. Drei Viertel der Patienten mit konventioneller Therapie war noch am Leben.
Wahrscheinlich renne ich mit diesem Artikel bei den meisten meiner Leserinnen und Leser offene Türen ein. Ich finde einen anderen Aspekt der „alternativen“ Therapien diskussionswürdig:
Mir fällt es als promoviertem Molekularbiologen und Proteinbiochemiker relativ einfach, den uns vorgeschlagenen alternativen Therapien argumentativ zu begegnen. Ich habe das Gefühl, ich erspare Titien dadurch seit der Diagnose und während ihrer Therapie eine Menge Unsicherheit und Zweifel.
Wie geht es wohl Patienten, die nicht in der Lage sind, medizinische gesichertes Wissen von alternativem Humbug zu unterscheiden? Die nicht wissen, ob sie ihrer Onkologin oder dem Freundeskreis oder der Webseite, die sie selbst auf Facebook „recherchiert“ haben trauen können?
Sich bei der Wahl der Therapie sicher zu fühlen trägt auch zur Lebensqualität Krebskranker bei.