Wenn Teile von Abbildungen in wissenschaftlichen Publikationen kopiert sind, dann ist das verdächtig. Wenn diese auch noch unterschiedliche Beschriftungen tragen und in unterschiedlichem Kontext gezeigt werden, dann ist das wissenschaftliches Fehlverhalten und Vorsatz kann unterstellt werden – ganz egal ob dies für die Kernaussage der Veröffentlichung relevant ist oder nicht.
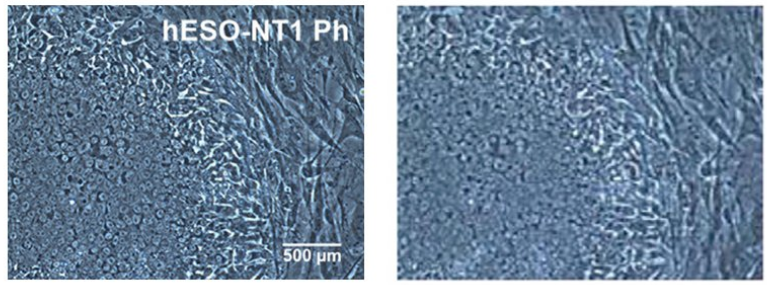
In einem in der letzten Woche in Cell publizierten Paper aus dem Labor von Shoukhrat Mitalipov fielen einer anonymen Wissenschaftlerin mehrere Unstimmigkeiten auf. Sie hat dies auf der Plattform Pubpeer dokumentiert, einer Seite auf der nach dem offiziellen Peer-Review wissenschaftliche Veröffentlichungen diskutiert und kritisiert werden können. Am auffälligsten dabei sind die Duplikationen von Bildausschnitten. Zusätzlich sind der anonymen Wissenschaftlerin noch weitere Unstimmigkeit in einer Abbildung aufgefallen, in der Genepxressionsdaten von vermeintlich unterschiedlichen biologischen Repliken verglichen werden.
Besonderere Brisanz wird diesem Fall dadurch verliehen, dass das Paper über die Herstellung embryonaler Stammzellen aus Körperzell-DNA und Eizellehülle in den Medien starke Resonanz fand. Und vor dem Hintergrund, dass ähnliche Versuche embryonale Stammzellen herzustellen, 2006 als Fälschung entlarvt wurden. Dementsprechend hoch ist auch aktuell die mediale Berichterstattung über die Unstimmigkeiten, zum Beispiel hier, hier oder hier.
Ich möchte hier versuchen, auf ein paar Fragen einzugehen, die vielleicht nicht detailliert genug in den großen Online-Blättern zur Sprache kommen.
Versehen oder Vergehen?
Mitalipov beschwichtigt in einem Interview in Nature und erklärt, es habe sich um ein Versehen gehandelt, das möglicherweise durch einen zu hastigen Begutachtungsprozess nicht entdeckt wurde. Meiner Meinung nach wurden die Bilder in Abbildung 6D und S5 vorsätzlich vertauscht und unterschiedlich beschriftet (siehe Abbildung hier oben). Wer Bilder am Mikroskop erstellt und speichert, sorgt dafür, dass diese eindeutig benannt und geordnet abgelegt werden. Wer eine veröffentlichungsreife Abbildung anfertigt, vergewissert sich mehrfach, dass das was man da sieht, das ist, was gezeigt werden soll. Wieso der Maßstab auf dem selben Bild einmal fehlt und einmal vorhanden ist, verstehe ich ebenfalls nicht.
Sind die Fehler relevant für die Kernaussage der Veröffentlichung?
Bei den vertauschten und zurechtgeschnittenen mikroskopischen Aufnahmen kann ich das nicht beurteilen. Die Fehler in Abbildung S6 hingegen sind jedoch sehr nahe dran. Ein Kernaspekt der Studie betrifft die Ähnlichkeit der hergestellten Stammzellen zu natürlichen embryonalen Stammzellen. Um das zu beurteilen, werden Genexpressionsmuster verglichen. Je besser diese korrelieren, desto mehr ähneln sich die Zellen. Man sieht das gut in Abbildung 7A. Generell werden solche Experimente in technischen sowie biologischen Replikaten durchgeführt (meistens drei), um die Stärke der Korrelation der Genexpressionsmuster besser beurteilen zu können. Das sind die Autoren in Abbildung S6 schuldig geblieben (hier unten).
Abbildung S6 aus dem Paper. Die Grafiken oben mitte und rechts sind identisch. Die Grafiken in der Mitte links und unten rechts sehen laut anonymem Gutachter bei PubPeer zu gut aus, um von biologischen Replikaten zu stammen.
Hat der Peer-Review Prozess versagt?
Nein. Versagt hat die Redaktion von Cell. Ich gehe eigentlich davon aus, dass die meisten Wissenschaftsmagazine – mit Sicherheit aber Cell – inzwischen Software besitzen um Fälschung in Abbildungen zu detektieren. Warum eine solche Software in diesem Fall nicht eingesetzt wurde, ist rätselhaft. Deutlich kritikwürdiger als der Reviewprozess ist also die mangelhafte redaktionelle Arbeit beim Elsevier – Flaggschiff.
War der Peer-Reveiw Prozess zu schnell?
Laut Angaben von Cell wurde das Paper am 30. April eingereicht, am 3. Mai überarbeitet und am selben Tag akzeptiert. Meiner Erfahrung nach spiegeln diese Daten nicht den Ablauf des tatsächlichen Begutachtungsprozesses wider. Normalerweise wird ein eingereichtes Manuskript als erstes redaktionell bewertet und entschieden, ob die Veröffentlichung überhaupt zu Gutachtern geschickt wird. Diese werden dann mit der Bitte kontaktiert, das Manuskript innerhalb von drei Wochen zurück zu schicken (nachzulesen auch hier). Danach kann eine Publikation entweder abgelehnt werden (sehr häufig) oder nach Bearbeitung der Anmerkungen der Gutachter durch die Autoren akzeptiert werden.
Um die offiziellen Begutachtungszeiten für Manuskripte niedrig zu halten (und die Ablehnungsraten hoch), greifen Journals zu einem Kniff: Papers, die stark überarbeitet werden müssen, werden abgelehnt. Allerdings wird den Autoren ermöglicht, das verbesserte Manuskript beim gleichen Magazin erneut einzureichen. Bei etwaiger Veröffentlichung gilt dann das zweite Einreichdatum, und das kann sehr nahe beim Datum liegen, an dem das Paper zur Veröffentlichung akzeptiert wurde.
Seltsam mutet daher die Antwort von Mitalipov auf die Frage an, warum solche Eile bei der Publikation der Daten bestand. Der Hauptautor der Studie nimmt Cell in Schutz und sagte dazu zu Nature, er wollte die Veröffentlichung beschleunigen, um die Ergebnisse bei einem Meeting im Juni zu präsentieren.
Seit wann müssen Daten publiziert sein, um bei einem Meeting präsentiert werden zu können? Und seit wann hat ein Autor einen solch großen Einfluss auf die Publikationspolitik eines Journals?
Autoren müssen bei der Veröffentlichung eines Papers immer etwaige Interessenkonflikte angeben. Wie werden wohl bei Cell Interessenkonflikte von Editoren behandelt?